Evolução e disseminação epidêmica do SARS-CoV-2 no Brasil
Publicado em: 05/08/2020, 18:00
Para ler o artigo na versão original (inglês) clique aqui
O Brasil atualmente tem uma das epidemias de SARS-CoV-2 de crescimento mais rápido no mundo. Devido aos dados disponíveis limitados, as avaliações do impacto das intervenções não farmacêuticas (INFs) sobre a disseminação do vírus continuam desafiadoras. Usando um modelo de transmissão orientada para a mobilidade, mostramos que as INFs reduziram o número de reprodução de >3 para 1-1,6 em São Paulo e no Rio de Janeiro. O sequenciamento de 427 novos genomas e a análise de um conjunto de dados genômicos geograficamente representativos identificaram >100 introduções internacionais de vírus no Brasil. Estimamos que a maioria (76%) das linhagens brasileiras tenha caído em três clados introduzidos da Europa entre 22 de fevereiro e 11 de março de 2020. Durante a fase inicial da epidemia, descobrimos que o SARS-CoV-2 se espalhava principalmente nas fronteiras locais e dentro do estado. Após esse período, apesar das reduções acentuadas nas viagens aéreas, estimamos várias exportações de grandes centros urbanos que coincidiram com um aumento de 25% na distância média percorrida em voos nacionais. Este estudo lança uma nova luz sobre a transmissão epidêmica e as trajetórias evolutivas das linhagens de SARS-CoV-2 no Brasil e fornece evidências de que as intervenções atuais permanecem insuficientes para manter a transmissão do vírus sob controle no país.
O coronavírus 2 da síndrome respiratória aguda grave (SARS-CoV-2) é um novo betacoronavírus com um genoma de 30 kb que foi relatado pela primeira vez em dezembro de 2019 em Wuhan, China (1, 2). O SARS-CoV-2 foi declarado uma emergência de saúde pública de interesse internacional em 30 de janeiro de 2020. Até 12 de julho 2020, a doença do coronavírus 2019 (COVID-19) causou mais de 12,5 milhões de casos e 561 mil mortes globalmente (3). O vírus pode ser classificado em duas linhagens filogenéticas principais, a saber A e B, que se espalharam de Wuhan antes de serem adotadas restrições rigorosas de viagens (4,5) e agora cocirculam pelo mundo (6).A taxa de fatalidade de casos de infecção por SARS-CoV-2 foi estimada entre 1,2 e 1,6% (7-9), com proporções substancialmente mais altas nas pessoas acima de 60 anos (8).Algumas estimativas sugerem que 18-56% da transmissão de SARS-CoV-2 é de indivíduos assintomáticos ou pré-sintomáticos (10-13), complicando as avaliações epidemiológicas e os esforços de saúde pública para conter a pandemia.
Desafios da avaliação em tempo real da transmissão
Embora as epidemias de SARS-CoV-2 em vários países, incluindo China, Itália e Espanha tenham sido controladas por intervenções não farmacêuticas (INFs) (3), o número de casos e mortes por SARS-CoV-2 no Brasil continua a aumenta (14) (Fig. 1A). Até 12 de julho de 2020, o Brasil já havia relatado 1.800.827 casos de SARS-CoV-2, o segundo maior número do mundo, e 70.398 mortes. Mais de um terço dos casos (34%) no Brasil estão concentrados na região sudeste, que inclui a cidade de São Paulo (Fig. 1B), a quarta maior conurbação do mundo, onde o primeiro caso na América Latina foi registrado em 25 de fevereiro de 2020 (15).Os ensaios de diagnóstico para detecção molecular do SARS-CoV-2 foram amplamente distribuídos pelos centros de referência regionais da rede nacional de laboratórios de saúde pública a partir de 21 de fevereiro de 2020 (16, 17).No entanto, vários fatores, incluindo atrasos nos relatórios, alterações na notificação e acesso heterogêneo a testes entre populações, ofuscam a avaliação em tempo real da transmissão de vírus usando contagens de casos de SARS-CoV-2 (15).Consequentemente, uma medida mais precisa da transmissão de SARS-CoV-2 no Brasil é fornecida pelas mortes relatadas por infecções respiratórias agudas graves (SARI), fornecidas pelo Sistema Único de Saúde (SUS) (18).Alterações na oportunidade de transmissão de SARS-CoV-2 estão fortemente associadas a alterações na mobilidade média (18-20) e geralmente podem ser medidas calculando-se o número efetivo de reprodução, R, definido como o número médio de infecções secundárias causadas por uma pessoa infectada. R >1 indica uma epidemia crescente, enquanto R <1 é necessário para obter uma diminuição na transmissão.
Utilizamos um modelo semimecanicista bayesiano (21, 22) para analisar estatísticas de mortalidade por SARI e dados de mobilidade humana para estimar mudanças diárias em R na cidade de São Paulo (12,2 milhões de habitantes) e na cidade do Rio de Janeiro (6,7 milhões de habitantes). ), as maiores metrópoles urbanas do Brasil (Fig. 1, C e D). As INFs no Brasil consistiram em fechamentos de escolas implementados entre 12 e 23 de março de 2020 nas 27 unidades/estados federais do país e fechamentos de lojas implementados entre 13 e 23 de março de 2020. Na cidade de São Paulo, as escolas começaram a fechar em 16 de março e as lojas fecharam quatro dias depois. No início da pandemia, encontramos R> 3 em São Paulo e Rio de Janeiro e, concomitantemente com a sincronização das INFs obrigatórios pelo estado, os valores de R caíram perto de 1.
Alterações no número de reprodução orientada pela mobilidade
A análise do número de reprodução após a implementação de INFs destaca várias características notáveis orientadas pela mobilidade. Houve um período imediatamente após as INFs, entre 21 e 31 de março de 2020, quando R foi consistentemente <1 na cidade de São Paulo (Fig. 1C). No entanto, após essa queda inicial, o valor de R para São Paulo aumentou para >1 e aumenta com o tempo, uma tendência associada ao aumento da mobilidade da população. Isso pode ser observado no índice de estações de trânsito do Google, que aumentou de 60% para -52%, e por uma diminuição no índice de isolamento social de 54% para 47%. Até 4 de maio de 2020, estimamos R = 1,3 (BCI 95%: 1,0-1,6) nas cidades de São Paulo e Rio de Janeiro (tabela S1). No entanto, observamos que houve casos nos 7 dias anteriores em que os intervalos de 95% confiáveis para R incluíram valores abaixo de 1, chamando a atenção para as flutuações e incertezas no número estimado de reprodução para ambas as cidades.
O compartilhamento inicial de sequências genômicas, incluindo o primeiro genoma SARS-CoV-2, Wuhan-Hu-1, lançado em 10 de janeiro (23), permitiu níveis globais sem precedentes de testes moleculares para um vírus emergente (24, 25).No entanto, apesar dos milhares de genomas de vírus depositados em bancos de dados de acesso público, há uma falta de estrutura de amostragem consistente e dados limitados do Brasil (26-28), o que dificulta reconstruções precisas do movimento e transmissão de vírus usando análises filogenéticas. Para investigar como o SARS-CoV-2 se estabeleceu no país e para quantificar o impacto das INFs na disseminação espaço-temporal do vírus, testamos um total de 26.732 amostras de laboratórios públicos e privados usando testes de PCR em tempo real e encontramos 7.944 (29% ) positivos para SARS-CoV-2. Em seguida, concentramos nossos esforços de sequenciamento na geração de um conjunto de dados genômico grande e espacialmente representativo com metadados selecionados, a fim de maximizar a associação entre o número de sequências e o número de casos de SARS-CoV-2 confirmados por estado.
Esforços de sequenciamento espacialmente representativos
Geramos 427 novos genomas de SARS-CoV-2 com cobertura >75% de genoma de amostras brasileiras coletadas entre 5 de março e 30 de abril de 2020 (figs. S1 a S3 e dados S1). Para cada estado, o tempo entre a data do primeiro caso relatado e a data de coleta da primeira sequência analisada nesse estado foi de apenas 4,5 dias em média (Fig. 2A). Para oito estados federais, os genomas foram obtidos a partir de amostras coletadas até 6 dias antes das primeiras notificações de casos. Os genomas gerados aqui foram coletados em 85 municípios em 18 das 27 unidades federais em todas as regiões do Brasil (Figura 2A e Figura S2). Os genomas sequenciados foram obtidos a partir de amostras coletadas em média 4 dias (mediana, intervalo: 0 a 29 dias) após o início dos sintomas e foram gerados em 3 laboratórios usando sequenciamento harmonizado e protocolos bioinformáticos (tabela S2). Quando incluímos 63 sequências adicionais disponíveis do Brasil depositadas no GISAID (29) (ver dados S1 e S2), descobrimos que o conjunto de dados é representativo da heterogeneidade espacial da epidemia brasileira. Especificamente, o número de genomas por estado correlaciona-se fortemente com os casos confirmados de SARS-CoV-2 com SARI e casos de SARI com etiologia desconhecida por estado (n = 490 sequências de 21 estados, correlação de Spearman, p = 0,83; Fig. 2A). Essa correlação variou de 0,70 a 0,83 ao considerar casos de SARI e mortes causadas por SARS-CoV-2, e casos de SARI e mortes por etiologia desconhecida (fig. S4). A maioria (n = 485/490) das sequências brasileiras pertence à linhagem B de SARS-CoV-2, com apenas 5 cepas pertencentes à linhagem A (2 do Amazonas, 1 do Rio Grande do Sul, 1 de Minas Gerais e 1 do Rio de Janeiro Além disso, foi utilizada uma avaliação in-silico da especificidade do teste diagnóstico para cepas brasileiras (n = 490) para identificar possíveis incompatibilidades em alguns ensaios direcionados às cepas brasileiras. Descobrimos que os iniciadores de direção do CDC chinês e da RT-qPCR direcionada à nucleoproteína da Universidade de Hong Kong podem ser menos apropriados para uso no Brasil do que outros ensaios de diagnóstico, para os quais poucas ou nenhuma incompatibilidade foi identificada (fig. S6 e tabela S3). O impacto dessas incompatibilidades na sensibilidade desses ensaios deve ser confirmado experimentalmente. Se a sensibilidade for afetada, o uso de testes RT-qPCR duplos que visam simultaneamente diferentes regiões genômicas pode ajudar na detecção de vírus com variantes nas regiões de ligação do iniciador ou da sonda.
Análises filogenéticas e introduções internacionais
Estimamos a probabilidade máxima e filogenias de relógio molecular para um conjunto de dados global com um total de 1.182 genomas amostrados de 24 de dezembro de 2019 a 30 de abril de 2020 (correlação de distância genética raiz a ponta com datas de amostragem, r2 = 0,53; Fig. 3A e fig. S7). Inferimos uma taxa evolutiva mediana de 1,13 x 10-3 (BCI 95%: 1,03-1,23 x 10-3) substituições por local por ano (s/s/y), usando um modelo coalescente de crescimento exponencial, equivalente a 33 alterações por ano, em média, no genoma do vírus. Isso está dentro da faixa de taxas evolutivas estimadas para outros coronavírus humanos (30-33). Estimamos a data do ancestral comum (TMRCA) da pandemia de SARS-CoV-2 em meados de novembro de 2019 (mediana = 19 de novembro de 2019, BCI 95%: 26 de outubro de 2019 a 6 de dezembro de 2019), em consonância com as descobertas recentes (34, 35).
A análise filogenética revelou que a maioria dos genomas brasileiros (76%, n = 370/490) se dividiu em três clados doravante denominados Clado 1 (n = 186/490, 38% das cepas brasileiras), Clado 2 (n = 166, 34%) e Clado 3 (n = 18/490, 4%; Fig. 3A e figs. S8 e S9), que estavam amplamente de acordo com aqueles identificados em uma análise filogenética usando 13.833 genomas globais. Os ancestrais comuns mais recentes dos três principais clados brasileiros (Clados 1 a 3) foram datados de 28 de fevereiro (21 de fevereiro a 4 de março de 2020) (Clado 1), 22 de fevereiro (17 a 24 de fevereiro de 2020) (Clado 2) a 11 Março (9 a 12 de março de 2020) (Clado 3) (Fig. 3A e fig. S10). Isso indica que a transmissão orientada pela comunidade já estava estabelecida no Brasil no início de março, sugerindo que as restrições internacionais de viagens iniciadas após esse período teriam um impacto limitado. O Clado 1 brasileiro é caracterizado por uma substituição nucleotídica da proteína spike (G25088T, numeração relativa à referência do Gen-Bank NC_045512.2) e circula predominantemente no estado de São Paulo (n = 159, 85,4%; figs. S9 e S11). O clado 2 é definido por duas substituições de nucleotídeos em ORF6 (T27299C) e nucleoproteína (T29148C); essa é a linhagem mais espacialmente difundida, com sequências de um total de 16 estados no Brasil. O Clado 3 está concentrado no estado do Ceará (n = 16, 89%) e cai em um conjunto global com sequências principalmente da Europa. Na região amazônica, onde a epidemia está se expandindo rapidamente (14, 22), encontramos evidências de diversas introduções nacionais e internacionais, com 37% (n = 7/19) das sequências dos estados do Pará e Amazonas agrupados no Clado 1 e 32% (n = 6/19) no Clado 2
As análises filogeográficas medidas no tempo revelaram pelo menos 102 (BCI 95%: 95-109) introduções internacionais de SARS-CoV-2 no Brasil (Fig. 3A e figs. S8 e S12). Isso representa uma subestimativa do número real de introduções, pois sequenciamos, em média, apenas 1 em cada 200 casos confirmados. A maioria dessas introduções estimadas foi direcionada para estados internacionalmente conectados (36), como São Paulo (36% de todas as importações), Minas Gerais (24%), Ceará (10%) e Rio de Janeiro (8%) (fig. S12). Avaliamos ainda a contribuição dos eventos de movimentação de linhagem internacional versus nacional ao longo do tempo (Fig. 3B). Na primeira fase da epidemia, encontramos um número crescente de introduções internacionais até 10 de março de 2020 (Fig. 2B). Dados limitados de histórico de viagens disponíveis (15) sugerem que esses casos iniciais foram predominantemente adquiridos da Itália (26%, n = 70 de 266 países com infecção sem ambiguidade) e dos EUA (28%, n = 76 de 266). Após essa fase inicial, descobrimos que o número estimado de importações internacionais diminuiu concomitantemente com o declínio no número de passageiros internacionais viajando para o Brasil (Fig. 3, B e C e S13). Por outro lado, apesar da queda no número de passageiros que viajam em voos nacionais (Fig. 3C), detectamos um aumento nos eventos de movimentação de linhagens de vírus entre as regiões brasileiras pelo menos até o início de abril de 2020.
Modelo da disseminação espaço-temporal no Brasil
Para entender melhor a disseminação do vírus por escalas espaço-temporais no Brasil, usamos um modelo filogeográfico contínuo que mapeia os nós filogenéticos para seus locais de origem inferidos (37) (Fig. 4). Distinguimos ramos que permanecem dentro de um estado versus aqueles que cruzam um estado para inferir a proporção de movimento do vírus observado dentro do estado versus entre estados.
Estimamos que, durante a primeira fase epidêmica, o SARS-CoV-2 se espalhe principalmente nas fronteiras locais e dentro do estado. Em contraste, a segunda fase foi caracterizada por eventos de movimento a longa distância e a ignição da epidemia fora da região sudeste do Brasil (Fig. 4A). Durante a epidemia, descobrimos que o movimento de linhagem de vírus dentro do estado foi, em média, 5,1 vezes mais frequente do que o movimento entre estados. Além disso, nossos dados sugerem que a disseminação do vírus dentro do estado e, em menor extensão, a disseminação de vírus entre estados, diminuiu após a implementação das INFs (Fig. 4B). No entanto, é útil observar que a amostragem mais limitada após 6 de abril de 2020 (ver fig. S2) diminuiu o movimento inferido da linhagem de vírus para o presente (Figs. 3B e 4B).
Curiosamente, descobrimos que o comprimento médio da rota percorrida pelos passageiros aumentou 25% durante a segunda fase da epidemia (Fig. 4C), apesar de uma redução concomitante no número de passageiros que voam no Brasil (Fig. 3C). O aumento na extensão média da rota após a implementação de INFs resulta de uma redução maior no número de passageiros aéreos que voam em viagens de menor distância em comparação com aqueles que praticam viagens de longa distância. Por exemplo, encontramos uma redução de 8,8 vezes no número de passageiros que voam nos segmentos de voo <1000 km, em comparação com uma redução de 4,4 vezes nos que voam >2000 km (fig. S15). Essas descobertas enfatizam os papéis da mobilidade dentro e entre estados como um fator-chave da disseminação local e inter-regional do vírus, com conurbações urbanas altamente povoadas e bem conectadas na região sudeste, atuando como principais fontes de exportação de vírus no país ( fig. S12).
Discussão
Fornecemos uma análise abrangente da disseminação do SARS-CoV-2 no Brasil, que mostra a importância de medidas comunitárias e nacionais para controlar a epidemia de COVID-19 no Brasil. Embora as INFs inicialmente tenham reduzido a transmissão e a disseminação de vírus, o aumento contínuo do número de casos e mortes no Brasil destaca a necessidade urgente de impedir a transmissão futura de vírus, implementando triagem diagnóstica rápida e acessível, rastreamento de contatos, quarentena de novos casos e coordenação social e física medidas de distanciamento em todo o país (38).Com o recente relaxamento das INFs no Brasil e em outros lugares, é necessária uma vigilância molecular, imunológica e genômica contínua para decisões baseadas em dados em tempo real. Nossa análise mostra como as mudanças na mobilidade podem afetar a transmissão global e local do SARS-CoV-2 e demonstra como a combinação de dados genômicos e de mobilidade pode complementar as abordagens tradicionais de vigilância.
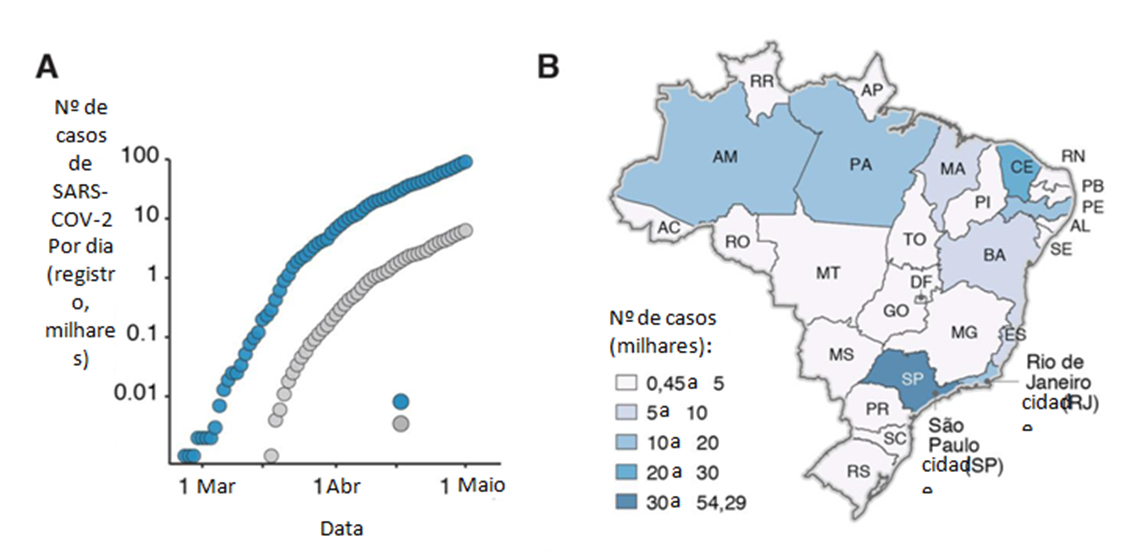
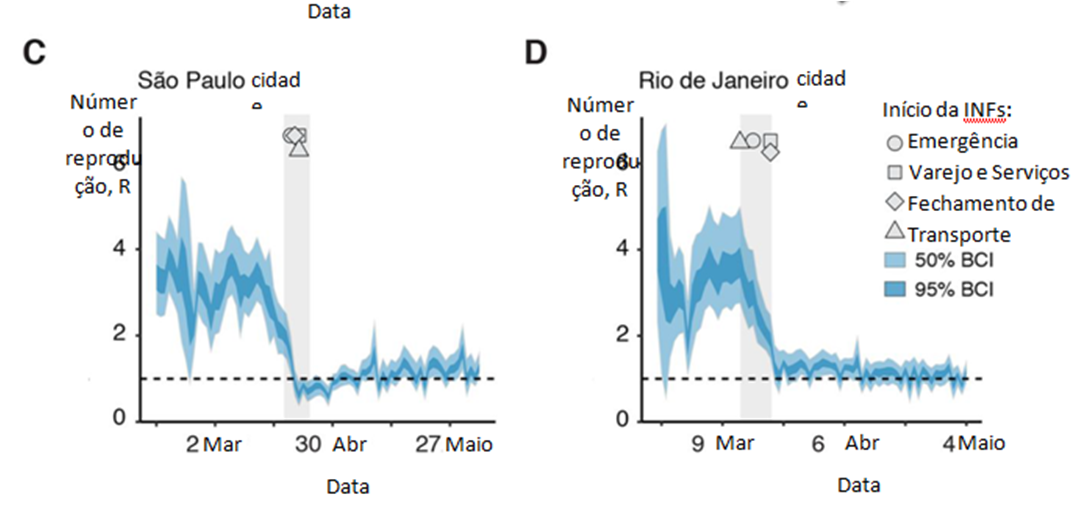
Fig. 1. Epidemiologia e disseminação epidêmica do SARS-CoV-2 no Brasil. (A) Número acumulado de casos notificados de SARS-CoV-2 (azul) e óbitos (cinza) no Brasil. (B) Os estados são coloridos de acordo com o número acumulado de casos confirmados até 30 de abril de 2020. (C e D) Número de reprodução (R) ao longo do tempo para as cidades de São Paulo (C) e Rio de Janeiro (D). R foram estimados usando uma abordagem bayesiana incorporando número diário de mortes e quatro variáveis relacionadas a dados de mobilidade (um índice de isolamento social da empresa brasileira de geolocalização InLoco e índices de mobilidade do Google para o tempo gasto em estações de trânsito, parques e a média entre alimentos e farmácias , varejo e recreacional e espaços de trabalho). A linha horizontal tracejada indica R = 1. A área cinza e os símbolos geométricos mostram os horários em que as intervenções das INFs foram implementadas. Intervalos credíveis bayesianos (BCIs, 50 e 95%) são mostrados como áreas sombreadas. Os códigos ISO 3166-1 de duas letras para as 27 unidades federais no Brasil são fornecidos em Informações Complementares.
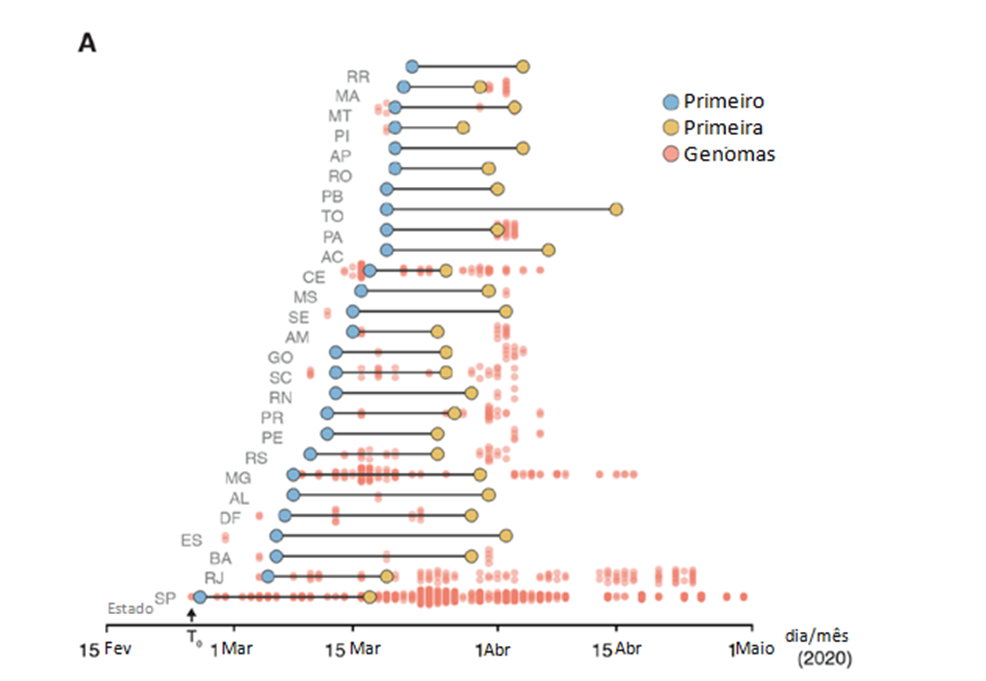
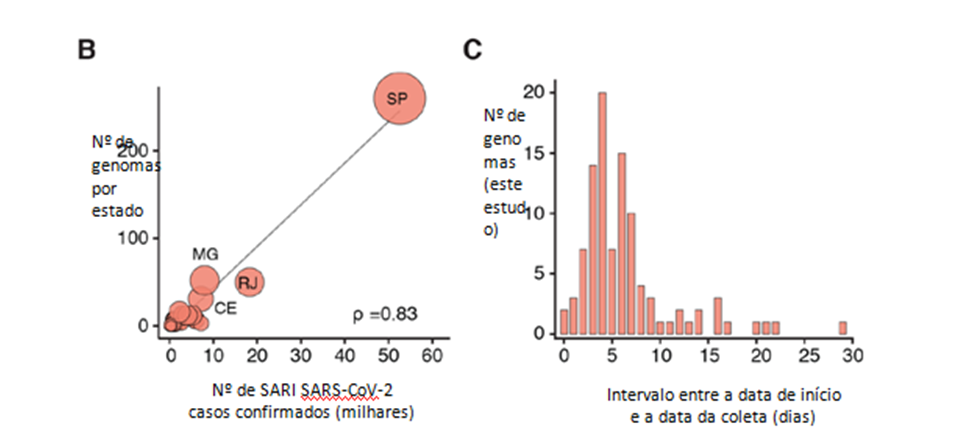
Fig. 2. Amostragem genômica espacialmente representativa. (A) Gráfico de halteres mostrando os intervalos de tempo entre a data da coleta dos genomas amostrados, a notificação dos primeiros casos e as primeiras mortes em cada estado. Linhas vermelhas indicam o atraso entre a data de coleta da primeira sequência do genoma e o primeiro caso relatado. Os códigos ISO 3166-1 de duas letras para as unidades federais brasileiras (ou estados) são fornecidos em Informações Complementares. (B) A correlação de Spearman (p) entre o número de casos confirmados de SARI por SARS-CoV-2 e SARI com etiologia desconhecida em relação ao número de sequências para cada um dos 21 estados brasileiros incluídos neste estudo (ver também fig. S4). Os tamanhos dos círculos são proporcionais ao número de sequências para cada unidade federal. (C) Intervalo entre a data de início dos sintomas e a data da coleta de amostras para as sequências geradas neste estudo.
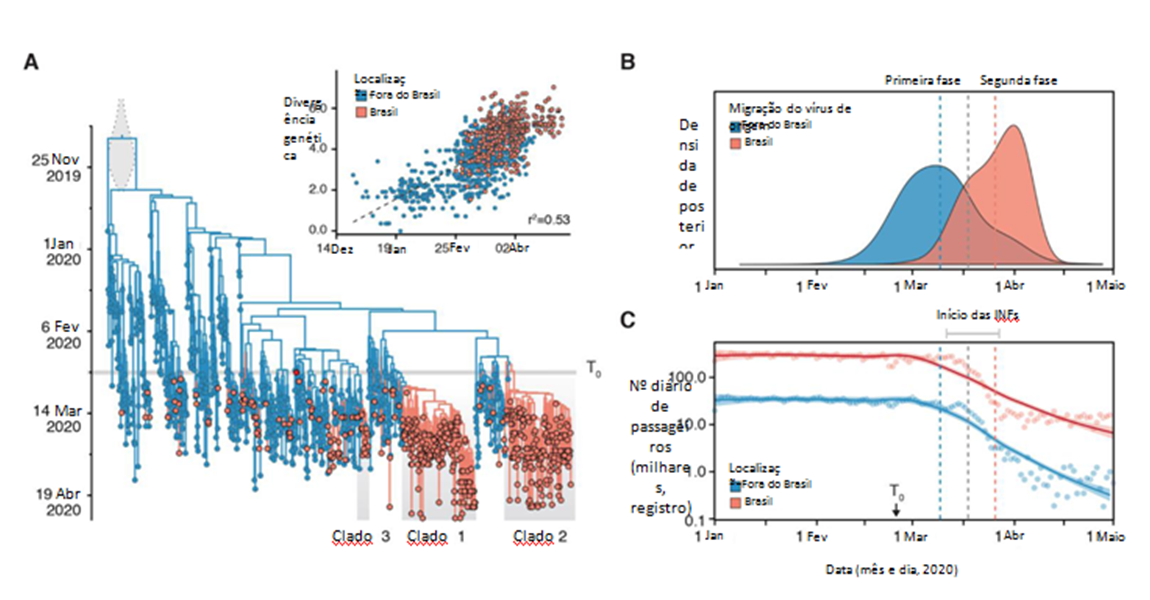
Fig. 3. Evolução e disseminação SARS-CoV-2 no Brasil (A) Filogenia de credibilidade máxima do clado resolvida no tempo de 1182 sequências SARS-CoV-2, 490 do Brasil (vermelho) e 692 do exterior (azul). Os maiores clados brasileiros são destacados por caixas cinzas (Clado 1, Clado 2 e Clado 3). A inserção do painel A mostra uma regressão raiz-a-ponta da divergência genética em relação às datas de coleta da amostra. (B) Dinâmica dos eventos de importação de f SARS-CoV-2 no Brasil. Datas de eventos de migração internacionais e nacionais (entre estados federais) foram estimadas a partir de genomas de vírus usando uma abordagem filogeográficas. A primeira fase foi dominada pela migração de vírus de fora do Brasil, enquanto a segunda fase é marcada pela disseminação do vírus no Brasil. As linhas verticais tracejadas correspondem à estimativa posterior média para eventos de migração de fora do Brasil (azul) e dentro do Brasil (vermelho). (C) Suavização localmente estimada do gráfico de dispersão do número diário de passageiros aéreos internacionais (azuis) e nacionais (vermelhos) no Brasil em 2020. To= data do primeiro caso relatado no Brasil (25 de fevereiro de 2020).
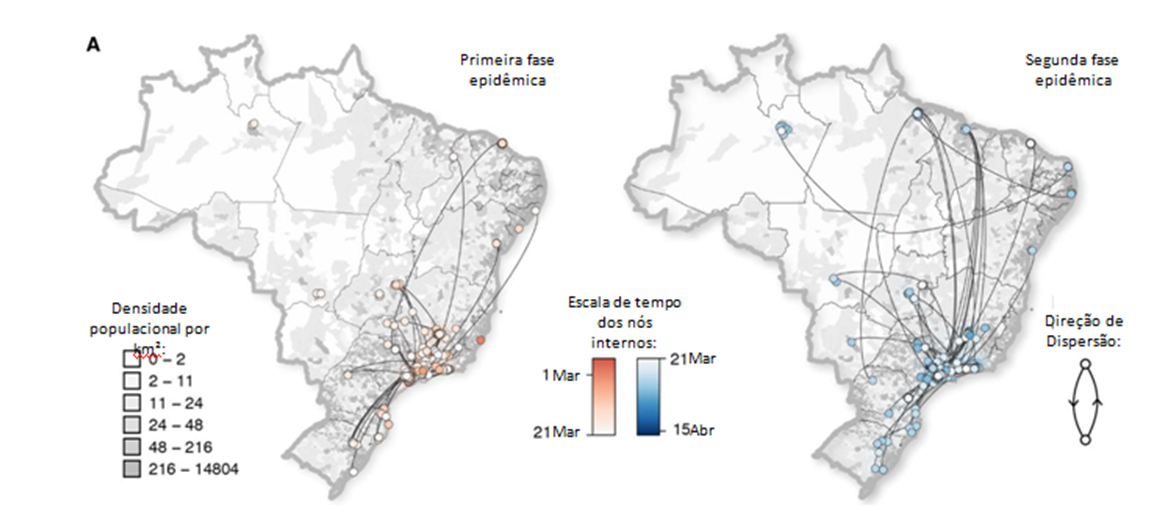
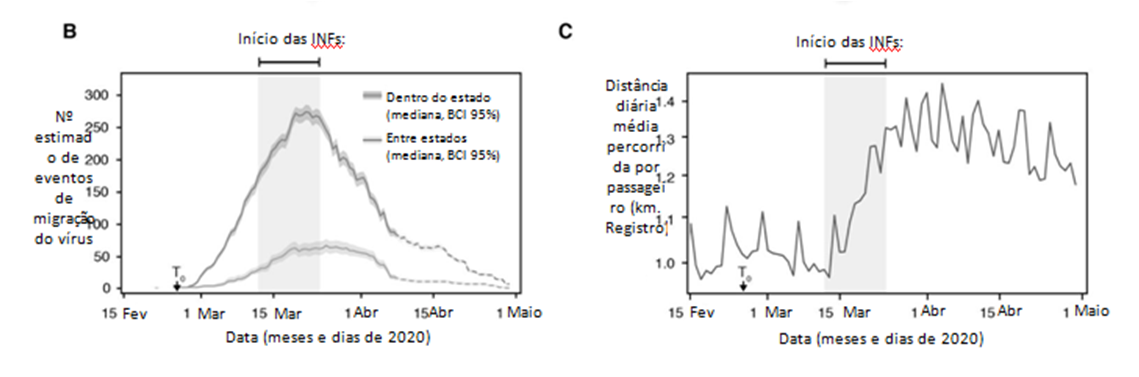
Fig. 4. Disseminação SARS-CoV-2 no Brasil (A) Reconstrução espaço-temporal da disseminação de conjuntos brasileiros de SARS-CoV-2 contendo >2 sequências durante a primeira fase epidêmica (esquerda) e a segunda fase epidêmica (direita) (fig. 3B). Os círculos representam nós da filogenia do MCC e são coloridos de acordo com o tempo de ocorrência inferido. As áreas sombreadas representam o intervalo de 80% da densidade posterior (HPD) mais alta e representam a incerteza das estimativas filogeográficas de cada nó. Linhas curvas sólidas denotam as relações entre os nós e a direcionalidade do movimento. As sequências pertencentes a conjuntos com <3 sequências também estão marcadas no map sem linhas conectando-as. A densidade populacional de fundo de cada município foi obtida no Instituto Brasileiro de Geografia (https://www.ibge.gov.br/).Ver fig. S14 para detalhes da disseminação do vírus na região Sudeste. (B) Número estimado de migrações de vírus dentro do estado (ou dentro de uma determinada unidade federal) e entre estados (ou entre unidades federais) ao longo do tempo. Linhas tracejadas indicam estimativas obtidas durante o período de amostragem limitada (fig. S2). (C) Distância média em quilômetros percorridos por passageiro aéreo por dia no Brasil. O número de passageiros aéreos diários é mostrado na Fig. 3B. Caixas cinza claro indicam as datas de início das INFs em todo o Brasil.
Science
Evolução e disseminação epidêmica da SARS-CoV-2 no Brasil
Darlan S. Candido, Ingra M. Claro, Jaqueline G. de Jesus, William M. Souza, Filipe R. R. Moreira, Simon Dellicour, Thomas A. Mellan, Louis du Plessis, Rafael H. M. Pereira, Flavia C. S. Sales, Erika R. Manuli, Julien Thézé, Luiz Almeida, Mariane T. Menezes, Carolina M. Voloch, Marcilio J. Fumagalli, Thaís M. Coletti, Camila A. M. da Silva, Mariana S. Ramundo, Mariene R. Amorim, Henrique H. Hoeltgebaum, Swapnil Mishra, Mandev S. Gill, Luiz M. Carvalho, Lewis F. Buss, Carlos A. Prete Jr, Jordan Ashworth, Helder I. Nakaya, Pedro S. Peixoto, Oliver J. Brady, Samuel M. Nicholls, Amilcar Tanuri, Átila D. Rossi, Carlos K.V. Braga, Alexandra L. Gerber, Ana Paula de C. Guimarães, Nelson Gaburo Jr, Cecila Salete Alencar, Alessandro C.S. Ferreira, Cristiano X. Lima, José Eduardo Levi, Celso Granato, Giulia M. Ferreira, Ronaldo S. Francisco Jr, Fabiana Granja, Marcia T. Garcia, Maria Luiza Moretti, Mauricio W. Perroud Jr, Terezinha M. P. P. Castiñeiras, Carolina S. Lazari, Sarah C. Hill, Andreza Aruska de Souza Santos, Camila L. Simeoni, Julia Forato, Andrei C. Sposito, Angelica Z. Schreiber, Magnun N. N. Santos, Camila Zolini de Sá, Renan P. Souza, Luciana C. Resende-Moreira, Mauro M. Teixeira, Josy Hubner, Patricia A. F. Leme, Rennan G Moreira, Maurício L. Nogueira, disseminação-UK Centre for Arbovirus Discovery, Diagnosis, Genomics and Epidemiology (CADDE) Genomic Network, Neil M Ferguson, Silvia F. Costa, José Luiz Proenca-Modena, Ana Tereza R. Vasconcelos, Samir Bhatt, Philippe Lemey, Chieh-Hsi Wu, Andrew Rambaut, Nick J. Loman, Renato S. Aguiar, Oliver G. Pybus, Ester C. Sabino and Nuno Rodrigues Faria
Publicado online em 23 de julho de 2020
AUTORES
Dalian S. Candido1,2*, Ingra M. Claro2,3*, Jaqueline G. de Jesus2,3*, William M. Souza4*, Filipe R. R. Moreira5*, Simon Dellicour6,7*, Thomas A. Mellan8*, Louis du Plessis1, Rafael H. M. Pereira9, Flavia C. S. Sales2,3, Erika R. Manuli2,3, Julien Theze10, Luiz Ahneida11, Mariane T. Menezes5, Carolina M. Voloch5, Marcilio J. Fumagalli4, Thais M. Coletti2,3, Camila A. M. da Silva2,3, Mariana S. Ramundo2,3, Mariene R. Amorim12, Henrique H. Hoeltgebaum13, Swapnil Mishra8, Mandev S. Gill7, Luiz M. Carvalho14, Lewis F. Buss2, Carlos A. Prete Jr15, Jordan Ashworth16, Helder I. Nakaya17, Pedro S. Peixoto18, Oliver J. Brady19,20, Samuel M. Nicholls21, Amilcar Tanuri5, Atila D. Rossi5, Carlos K.V. Braga9, Alexandra L. Gerber11, Ana Paula de C. Guimaries11, Nelson Gaburo Jr22, Cecila Salete Alencar23, Alessandro C.S. Ferreira 24, Cristiano X. Lima25,26, Jose Eduardo Levi27, Celso Granato28, Giulia M. Ferreira29, Ronaldo S. Francisco Jr11, Fabiana Granja12,30, Marcia T. Garcia31, Maria Luiza Moretti31, Mauricio W. Perroud Jr32, Terezinha M. P. P. Castirieiras33, Carolina S. Lazari34, Sarah C. Hill1,35, Andreza Aruska de Souza Santos36, Camila L. Simeoni12, Julia Forato12, Andrei C. Sposito37, Angelica Z. Schreiber38, Magnun N. N. Santos38, Camila Zolini de Si39, Renan P. Souza39, Luciana C. Resende-Moreira40, Mauro M. Teixeira 41, Josy Hubner42, Patricia A. F. Leme43, Rennan G Moreira44, Mauricio L. Nogueira45, disseminação-UK Centre for Arbovirus Discovery, Diagnosis, Genomics and Epidemiology (CADDE) Genomic Network, Neil M Ferguson8, Silvia F. Costa 2,3, Jose Luiz Proenca-Modena 12, Ana Tereza R. Vasconcelos11, Samir Mate, Philippe Lemey7, Chieh-Hsi Wu46, Andrew Rambaut47, Nick J. Loman21, Renato S. Aguiar39, Oliver G. Pybusl, Ester C. Sabino2,3†, Nuno Rodrigues Faria1,2,8†
1Department of Zoology, University of Oxford, Oxford, UK. 2lnstituto de Medicina Tropical, Faculdade de Medicina da Universidade de São Paulo, São Paulo, disseminação. 3Departamento de Moléstias Infecciosas e Parasitarias, Faculdade de Medicina da Universidade de São Paulo, São Paulo, disseminação. 4Centro de Pesquisa em Virologia, Faculdade de Medicina de Ribeirão Preto, Ribeirão Preto, disseminação. 5Departamento de Genética, Instituto de Biologia, Universidade Federal do Rio de Janeiro, Rio de Janeiro, disseminação. 6Spatial Epidemiology Lab, Universite Libre de Bruxelles, Brussels, Belgium. 7 Department of Microbiology, Immunology and Transplantation, Rega Institute, KU Leuven, Leuven, Belgium. 8MRC Centre for Global Infectious Disease Analysis, J-IDEA, Imperial College London, London, UK. 9Institute for Applied Economic Research, Brasília, disseminação. 10Universite Clermont Auvergne, INRAE, VetAgro Sup, UMR EPIA, Saint-Genes-Champanelle, France.11Laboratório de Bioinformática, Laboratório Nacional de Computação Cientifica, Petrópolis, Brazi1.12Departamento de Genética, Evolução, Microbiologia e Imunologia, Instituto de Biologia and Experimental Medicine Research Cluster (EMRC), Universidade Estadual de Campinas, Campinas, Brazi1.13Department of Mathematics, Imperial College London, London, UK.14Escola de Matemática Aplicada (EMAp), Fundação Getúlio Vargas, Rio de Janeiro, disseminação. 15Department of Electronic Systems Engineering, University of São Paulo, São Paulo, disseminação. 16Usher Institute, University of Edinburgh, Edinburgh, UK.17Department of Clinical and Toxicological Analyses, School of Pharmaceutical Sciences, University of São Paulo, São Paulo, disseminação.18Departamento de Matemática Aplicada, Instituto de Matemática e Estatística, Universidade de São Paulo, São Paulo, disseminação. 19Department of Infectious Disease Epidemiology, Faculty of Epidemiology and Population Health, London School of Hygiene & Tropical Medicine, London, UK. 20Centre for the Mathematical Modelling of Infectious Diseases, London School of Hygiene & Tropical Medicine, London, UK. 21Institute for Microbiology and Infection, University of Birmingham, Birmingham, UK. 22DB Diagn6sticos do Brasil, São Paulo, disseminação. 23LIM 03 Laboratorio de Medicina Laboratorial, Hospital das Clinicas Faculdade de Medicina da Universidade de São Paulo, São Paulo, disseminação. 24lnstituto Hermes Pardini, Belo Horizonte, disseminação. 25Departamento de Cirurgia, Faculdade de Medicina, Universidade Federal de Minas Gerais, Belo Horizonte, disseminação. 26Simile Instituto de Imunologia Aplicada Ltda, Belo Horizonte, disseminação. 27Laboratório DASA, São Paulo, disseminação. 28Laboratório Fleury, São Paulo, disseminação. 29Laboratorio de Virologia, Instituto de Ciencias Biomédicas, Universidade Federal de Uberlândia, Uberlândia, disseminação. 30Centro de Estudos da Biodiversidade, Universidade Federal de Roraima, Boa Vista, disseminação. 31Divisão de Doenças Infecciosas, Faculdade de Ciencias Medicas, Universidade Estadual de Campinas, Campinas, disseminação. 32Hospital Estadual Sumaré, Universidade Estadual de Campinas, Campinas, disseminação. 33Departamento de Doenças Infecciosas e Parasitarias, Faculdade de Medicina, Universidade Federal do Rio de Janeiro, Rio de Janeiro, disseminação. 34Divisão de Laboratório Central do Hospital das Clinicas, da Faculdade de Medicina da Universidade de São Paulo, São Paulo, disseminação. 35Department of Pathobiology and Population Sciences, Royal Veterinary College, Hatfield, UK. 36University of Oxford, Latin American Centre, Oxford School of Global and Area Studies, Oxford, UK. 37Departamento de Clinica Medica, Faculdade de Ciencias Medicas, Universidade Estadual de Campinas, Campinas, disseminação. 38Departamento de Patologia Clinica, Faculdade de Ciencias Medicas, Universidade Estadual de Campinas, Campinas, disseminação. 39Departamento de Genética, Ecologia e Evolução, Instituto de Ciencias Biológicas, Universidade Federal de Minas Gerais, Belo Horizonte, disseminação. 40Departamento de Botânica, Instituto de Ciencias Biológicas, Universidade Federal de Minas Gerais, Belo Horizonte, disseminação. 41Departamento de Bioquímica e Imunologia, Universidade Federal de Minas Gerais, Belo Horizonte, disseminação. 42Departamento de Biologia Celular, Instituto de Ciencias Biológicas, Universidade Federal de Minas Gerais, Belo Horizonte, disseminação.43Centro de Saúde da Comunidade, Universidade Estadual de Campinas, Campinas, disseminação.44Centro de Laboratórios Multiusuários, Instituto de Ciencias Biológicas, Universidade Federal de Minas Gerais, Belo Horizonte, disseminação. 45Laboratorio de Pesquisas em Virologia, Faculdade de Medicina de São Jose do Rio Preto, São Jose do Rio Preto, São Paulo, disseminação. 46Mathematical Sciences, University of Southampton, Southampton, UK. 47lnstitute of Evolutionary Biology, University of Edinburgh, Edinburgh, UK.
*Estes autores contribuíram igualmente para este trabalho.
†Autor correspondente. E-mail: sabinoec@usp.br (E.C.S.); nuno.faria@zoo.ox.ac.uk (N.R.F.)
FERRAMENTAS DO ARTIGO
http://science.sciencemag.org/content/early/2020/07/22/science.abd2161
MATERIAIS COMPLEMENTARES
http://science.sciencemag.org/content/suppl/2020/07/22/science.abd2161.DC1
REFERÊNCIAS
Este artigo cita 58 artigos, 12 dos quais é possível acessar gratuitamente http://science.sciencemag.org/content/early/2020/07/22/science.abd2161#BIBL
AUTORIZAÇÕES
http://www.sciencemag.org/help/reprints-and-permissions
O uso deste artigo está sujeito aos Termos de Serviço





