Como a nova variante do SARS-CoV-2 impacta nos testes moleculares já existentes?
Publicado em: 29/12/2020, 19:07

Os vírus são conhecidos por se replicarem de forma muito rápida, e devido à falta de um mecanismo de conferência e correção dos erros causados pela sua enzima de replicação, ocorrem muitas falhas durante seu ciclo. Essas alterações podem ser benéficas para o vírus, permitindo a seleção dessa cepa alterada.
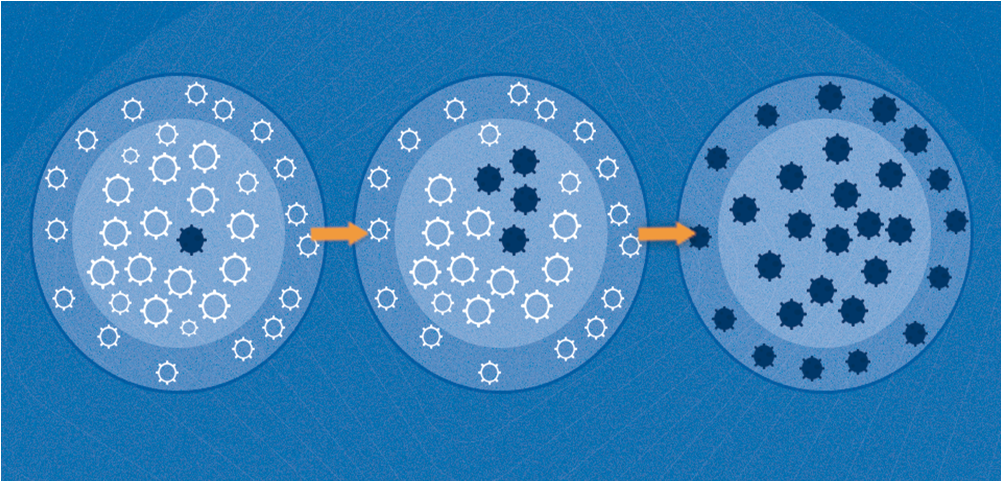
Figura 1: Representação da seleção de uma nova variante viral.
Recentemente foi reportada a presença de uma nova cepa variante de SARS-CoV-2, referida como "SARS-CoV-2 VOC 202012/01" ou "B.1.1.7.", que contém uma série de mutações. Essa variante descrita no Reino Unido (UK) tornou-se altamente prevalente em Londres e no sudeste da Inglaterra.
A B.1.17 possui uma mutação no domínio de ligação ao receptor (RBD) da proteína Spike na posição 501, onde o aminoácido asparagina (N) foi substituído por tirosina (Y). A abreviação para essa mutação é N501Y, podendo também ser nomeada como S: N501Y para especificar o local onde ocorreu a modificação.
Como descrito anteriormente, essa variante não possui apenas essa alteração genética, sendo também encontradas outras 14 mutações não sinônimas (quando a modificação causa alteração do AA codificado), 6 sinônimas (não há alteração do aminoácido [AA]) e 3 deleções. Entre essas alterações encontram-se:
— Deleção 69/70: essa deleção dupla ocorreu espontaneamente muitas vezes e provavelmente levou a uma mudança conformacional da proteína Spike.
— P681H: próximo ao local de clivagem da furina S1 / S2, um local com alta variabilidade em coronavírus. Essa mutação também surgiu espontaneamente várias vezes.
— Códon de parada ORF8 (Q27stop): Essa mutação não está na proteína Spike, mas em um gene diferente (ORF8), cuja função é desconhecida. Mutações semelhantes ocorreram no passado. Em Cingapura, uma cepa com esse tipo de mutação surgiu e desapareceu.
Em 18 de dezembro de 2020, o governo sul-africano anunciou que também presenciara o surgimento de uma nova cepa em um cenário semelhante ao do Reino Unido. A variante sul-africana também tem a mutação N501Y e várias outras mutações, mas surgiu de forma completamente independente da cepa do Reino Unido e não está relacionada a ela.
Essa nova mutação tem sido associada à hipótese de maior potencial de transmissão além de preocupar o mundo em relação aos aspectos epidemiológicos, à patogenicidade, ao impacto sobre a resposta vacinal e na detecção viral por testes diagnósticos de biologia molecular. No Brasil ainda não há relatos da circulação dessa nova variante contendo essa mutação.
Como a sequência do gene, que codifica para a proteína Spike, pode ser um dos alvos para detecção do genoma do vírus em kits de diagnóstico molecular, é importante esclarecer que o DB molecular utiliza sempre em seus ensaios mais de um “alvo” nas reações de PCR, como por exemplo sequência dos genes N (proteína de nucleocapsídeo), gene E (proteína de envelope), RdRP e ORF1ad evitando assim a possibilidade de ocorrência de testes falso negativos.
Esclarecemos ainda que o DB Molecular mantém um estado de constante vigilância para a presença de resultados inesperados, bem como realiza o encaminhamento das possíveis amostras atípicas para o sequenciamento de genoma completo viral em parceria com o Laboratório de Genética e microorganismos do Instituto de Medicina Tropical da USP coordenado pela Profª Drª Ester Sabino.
DB Molecular: Segurança, Qualidade e Inovação estão no nosso DNA.
Para ler a matéria completa do CDC (Centers for Disease Control and Prevention), clique aqui
Referências:
https://www.cdc.gov/mmwr/Novel_Coronavirus_Reports.html
Mutation rates among RNA viruses. Drake JW, Holland JJ. Proc Natl Acad Sci U S A. 1999 Nov 23; 96(24):13910-3.
RNA virus mutations and fitness for survival. Domingo E, Holland JJ. AnnuRev Microbiol. 1997; 51():151-78.





